
聚类
聚类和分类的区别:
分类其实是从特定的数据中挖掘模式,作出判断的过程。比如Gmail邮箱里有垃圾邮件分类器,一开始的时候可能什么都不过滤,在日常使用过程中,我人工对于每一封邮件点选“垃圾”或“不是垃圾”,过一段时间,Gmail就体现出一定的智能,能够自动过滤掉一些垃圾邮件了。这是因为在点选的过程中,其实是给每一条邮件打了一个“标签”,这个标签只有两个值,要么是“垃圾”,要么“不是垃圾”,Gmail就会不断研究哪些特点的邮件是垃圾,哪些特点的不是垃圾,形成一些判别的模式,这样当一封信的邮件到来,就可以自动把邮件分到“垃圾”和“不是垃圾”这两个我们人工设定的分类的其中一个。 聚类的的目的也是把数据分类,但是事先我是不知道如何去分的,完全是算法自己来判断各条数据之间的相似性,相似的就放在一起。在聚类的结论出来之前,我完全不知道每一类有什么特点,一定要根据聚类的结果通过人的经验来分析,看看聚成的这一类大概有什么特点。
聚类问题
所谓聚类问题,就是给定一个元素集合D,其中每个元素具有n个可观察属性,使用某种算法将D划分成k个子集,要求每个子集内部的元素之间相异度尽可能低,而不同子集的元素相异度尽可能高。其中每个子集叫做一个簇。
K-means聚类算法
聚类算法属于非监督学习的一种,聚类算法有很多种,大概有几十种聚类算法。 K-means是聚类算法中的最常用的一种,它运算速度快,但是只能应用于连续的数据。 K-means的算法的计算过程如下: 1,需要一个k值 来指定我们首先希望簇的个数(或者说首先定多少个中心点) 2,从数据集中随机选择k个数据点作为质心 3,计算每个点离质心的距离,选择最近的质心成一簇 4,每个簇算出新的质心 5,如果新的质心与老质心的距离小于某个阈值,则认为聚类达到期望 6,重复3-5步骤
"距离"的概念
K-means中的距离,是个相似度或者相异度的概念,根据数据集的特征(比如标量、向量、枚举分类等)决定如何计算距离。 常用的距离有: 1,欧几里德距离: 对于数值型、标量的数据,就应该用欧氏距离。 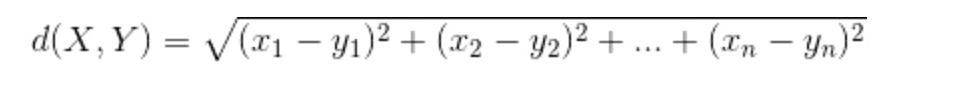 选择欧几里德距离计算距离,如果数据集的单位不一致,就会有问题。 所以要进行数据的标准化将数据按比例缩放,使之落入一个小的特定区间。去除数据的单位限制,将其转化纯数值型数据 标准化方法最常用的有两种: min-max标准化(离差标准化):对原始数据进行线性变换,是结果落到【0,1】区间,转换方法为 X'=(X-min)/(max-min),其中max为样本数据最大值,min为样本数据最小值。 z-score标准化(标准差标准化):处理后的数据符合标准正态分布(均值为0,方差为1),转换公式:X减去均值,再除以标准差 2,余弦相似度(向量夹角): 对于向量型的数据,使用标量型的距离公式就不合适了,一种流行的方法是计算两个向量的向量夹角。
选择欧几里德距离计算距离,如果数据集的单位不一致,就会有问题。 所以要进行数据的标准化将数据按比例缩放,使之落入一个小的特定区间。去除数据的单位限制,将其转化纯数值型数据 标准化方法最常用的有两种: min-max标准化(离差标准化):对原始数据进行线性变换,是结果落到【0,1】区间,转换方法为 X'=(X-min)/(max-min),其中max为样本数据最大值,min为样本数据最小值。 z-score标准化(标准差标准化):处理后的数据符合标准正态分布(均值为0,方差为1),转换公式:X减去均值,再除以标准差 2,余弦相似度(向量夹角): 对于向量型的数据,使用标量型的距离公式就不合适了,一种流行的方法是计算两个向量的向量夹角。  我们高中学过 两个向量的余弦是两个向量的点乘除以两个向量模长的乘积。 (两个向量的点乘也可以用向量矩阵X的转秩乘向量矩阵Y来表示)
我们高中学过 两个向量的余弦是两个向量的点乘除以两个向量模长的乘积。 (两个向量的点乘也可以用向量矩阵X的转秩乘向量矩阵Y来表示)  这里要注意,余弦度量度量的不是两者的相异度,而是相似度,所以我们应该使用他的反函数,即acos。 3,Jaccard: 就是交集除以并集 |S ∩ T|/|S ∪ T| 还有一些其他的距离计算方式:切比雪夫距离、曼哈顿距离、马哈拉诺比斯距离、调整后的余弦相似度等等
这里要注意,余弦度量度量的不是两者的相异度,而是相似度,所以我们应该使用他的反函数,即acos。 3,Jaccard: 就是交集除以并集 |S ∩ T|/|S ∪ T| 还有一些其他的距离计算方式:切比雪夫距离、曼哈顿距离、马哈拉诺比斯距离、调整后的余弦相似度等等
demo
from collections import defaultdict
from random import uniform
# from math import acos
from numpy import *
def point_avg(points):
"""
Accepts a list of points, each with the same number of dimensions.
NB. points can have more dimensions than 2
Returns a new point which is the center of all the points.
"""
dimensions = len(points[0])
new_center = []
for dimension in xrange(dimensions):
dim_sum = 0 # dimension sum
for p in points:
dim_sum += p[dimension]
# average of each dimension
new_center.append(dim_sum / float(len(points)))
return new_center
def update_centers(data_set, assignments):
"""
Accepts a dataset and a list of assignments; the indexes
of both lists correspond to each other.
Compute the center for each of the assigned groups.
Return `k` centers where `k` is the number of unique assignments.
"""
new_means = defaultdict(list)
centers = []
for assignment, point in zip(assignments, data_set):
new_means[assignment].append(point)
for points in new_means.itervalues():
centers.append(point_avg(points))
return centers
def assign_points(data_points, centers):
"""
Given a data set and a list of points betweeen other points,
assign each point to an index that corresponds to the index
of the center point on it's proximity to that point.
Return a an array of indexes of centers that correspond to
an index in the data set; that is, if there are N points
in `data_set` the list we return will have N elements. Also
If there are Y points in `centers` there will be Y unique
possible values within the returned list.
"""
assignments = []
for point in data_points:
shortest = 10 # positive infinity
shortest_index = 0
for i in xrange(len(centers)):
val = distance(point, centers[i])
if val < shortest:
shortest = val
shortest_index = i
assignments.append(shortest_index)
return assignments
def distance(a, b):
"""
"""
# d
dimensions = len(a)
_sum = 0
for dimension in xrange(dimensions):
difference_sq = (a[dimension] - b[dimension]) ** 2
_sum += difference_sq
return sqrt(_sum)
# acos
# difference_sq = dot(a,b)/(linalg.norm(a)*linalg.norm(b))
# return acos(difference_sq)
def generate_k(data_set, k):
"""
Given `data_set`, which is an array of arrays,
find the minimum and maximum for each coordinate, a range.
Generate `k` random points between the ranges.
Return an array of the random points within the ranges.
"""
centers = []
dimensions = len(data_set[0])
min_max = defaultdict(int)
for point in data_set:
for i in xrange(dimensions):
val = point[i]
min_key = 'min_%d' % i
max_key = 'max_%d' % i
if min_key not in min_max or val < min_max[min_key]:
min_max[min_key] = val
if max_key not in min_max or val > min_max[max_key]:
min_max[max_key] = val
for _k in xrange(k):
rand_point = []
for i in xrange(dimensions):
min_val = min_max['min_%d' % i]
max_val = min_max['max_%d' % i]
rand_point.append(uniform(min_val, max_val))
centers.append(rand_point)
return centers
def k_means(dataset, k):
k_points = generate_k(dataset, k)
assignments = assign_points(dataset, k_points)
old_assignments = None
while assignments != old_assignments:
new_centers = update_centers(dataset, assignments)
old_assignments = assignments
assignments = assign_points(dataset, new_centers)
return assignments